

蛋白组学一、肽段长度分布
每种质谱仪都有自身的测量范围,因此可鉴定到的肽段也有一定的长度限制。肽段过长或过短都无法在质谱仪中被检测到。如果鉴定结果中肽段普遍过低或普遍过高,则可能是蛋白酶选用不恰当。 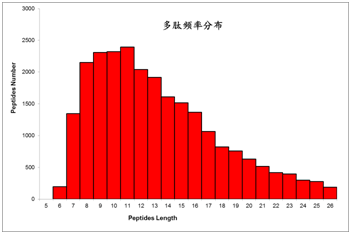
二、定量比值直方分布分析
样品定量结果中大部分蛋白质Ratio比值应接近1。运用频数分布直方图对Label-free定量数据进行分析,两组样本定量比值(FC=fold change)取Log2对数,见图5-2,纵坐标为蛋白质数,横坐标分别为两组样本定量比值(FC=fold change)的Log2对数值。
利用T检验分析出两样本间显著差异表达的蛋白后,以两组样本定量比值(FC=fold change)求取log10对数为横坐标,以T检验显著性检验P值的负对数-log10(P-value)为纵坐标,即可得火山图(Volcano Plot)(图5-3),利用一定的筛选条件(如大于2.0倍变化或/且P≤0.05),可以筛选出显著差异表达的蛋白,进行后续研究。火山图中的红色、绿色圆点表示具有显著性差异的蛋白质(满足FC≤0.5-绿色,or FC≥2.0-红色),灰色圆点为无变化的蛋白质(0.5<FC<2.0)。
聚类分析是模式识别和数据挖掘中普遍使用的一种方法,是基于数据的知识发现的有效方法。聚类分析不需要任何先验领域知识,它根据数学特征提取分类标准,对数据进行分类,发现对象之间的相似度。我们利用多样品间表达模式聚类分析观察不同蛋白在不同样品间比较时的上调、下调情况。图5-4是本项目中的差异蛋白聚类分析,图中的每一行代表一个蛋白,每一列为一个样本/重复,不同颜色表示不同的表达量(作图时对定量值进行log10取值,并进行中位校正)。
五、GO分析
Gene Ontology(简称GO)是生物信息领域中一个极为重要的方法和工具,通过建立一套具有动态形式的控制字集(controlled vocabulary),来解释真核基因及蛋白质在细胞内所扮演的角色,从而来全面描述生物体中基因和基因产物的属性。GO总共有三个本体(Ontology),分别描述基因的分子功能(Molecular Function)、所处的细胞位置(Cellular Component)、参与的生物过程(Biological Process)。 更详细信息见http://www.geneontology.org。 我们针对鉴定出的差异蛋白进行GO功能注释分析,给出的结果为差异蛋白的GO功能注释(图6-1)。
六、差异蛋白GO富集
GO功能显著性富集分析给出与所有鉴定到的蛋白质背景相比,差异蛋白质中显著富集的GO功能条目,从而给出差异蛋白质与哪些生物学功能显著相关。该分析首先把所有差异蛋白质向Gene Ontology数据库(http://www.geneontology.org/)的各个term映射,计算每个term的蛋白质数目,然后应用超几何检验,找出与所有蛋白质背景相比,在差异蛋白质中显著富集的GO条目。其计算公式为:
其中N为所有蛋白中具有GO 注释信息的蛋白数目,n为N中差异蛋白的数目,M为所有蛋白中注释到某个GO 条目的蛋白数目,m为注释到某个GO 条目的差异蛋白数目。计算得到P-value值,以P-value≤0.05为阈值,满足此条件的GO term定义为在差异蛋白质中显著富集的GO term。通过GO显著性分析能确定差异蛋白行使的主要生物学功能。
七、Pathway代谢通路注释 在生物体内,不同蛋白相互协调行使其生物学行为,基于Pathway的分析有助于更进一步了解其生物学功能。KEGG是有关Pathway的主要公共数据库(Kanehisa,2008),通过Pathway分析能确定蛋白质参与的最主要生化代谢途径和信号转导途径。我们针对鉴定出的差异蛋白进行kegg功能注释分析,给出的结果为差异蛋白的kegg功能注释。
八、差异蛋白pathway富集 Pathway显著性富集分析方法同GO功能富集分析,是以KEGG Pathway为单位,应用超几何检验,找出与所有鉴定到蛋白背景相比,在差异蛋白中显著性富集的Pathway。通过Pathway显著性富集能确定差异蛋白参与的最主要生化代谢途径和信号转导途径。 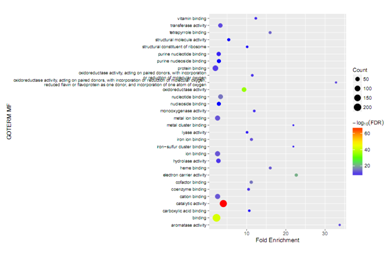
九、COG分析
COG(Cluster of Orthologous Groups of proteins 蛋白相邻类的聚簇)是对蛋白质进行直系同源分类的数据库。构成每个COG的蛋白都是被假定为来自于一个祖先蛋白,并且因此或者是orthologs或者是paralogs。Orthologs是指来自于不同物种的由垂直家系(物种形成)进化而来的蛋白,并且典型的保留与原始蛋白有相同的功能。Paralogs是那些在一定物种中的来源于基因复制的蛋白,可能会进化出新的与原来有关的功能。我们将鉴定到的差异蛋白蛋白和COG/KOG数据库(原核生物使用COG数据库,真核生物使用KOG数据库)进行比对,预测这些蛋白可能的功能并对其做功能分类统计。
|